10.31047/1668.298x.v40.n1.39619
COMUNICACIÓN
Protocolo para automatizar la descarga de datos climáticos desde la nube y generar indicadores biometeorológicos para el monitoreo epidemiológico de cultivos
P. Paccioretti
F Giannini Kurina
F Suarez
M. Scavuzzo
V. M. Alemandri
B. Gómez Montenegro
M. Balzarini
1 Paccioretti, P (ORCID: 0000-0002-1267-6361): Universidad Nacional de Córdoba, Facultad de Ciencias Agropecuarias e Instituto de Altos Estudios Espaciales “Mario Gulich”, Comisión Nacional de Actividades Espaciales, Falda del Cañete, Argentina. Giannini-Kurina, F (ORCID: 0000-0001 6763-7792): Aarhus Universitet, Institut for Agrookologi, Jorn&r Sektioner, Dinamarca. Suarez, F (ORCID: 0000-0001-6763-7792) y Balzarini, M. (ORCID: 0000-0002-4858-4637): Universidad Nacional de Córdoba, Facultad de Ciencias Agropecuarias y Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), Unidad de Fitopatología y Modelización Agrícola (UFYMA), Córdoba, Argentina. Scavuzzo, M., (ORCID: /0000-0003-0905-6361): Comisión Nacional de Actividades Espaciales, Universidad Nacional de Córdoba, Instituto de Altos Estudios Espaciales “Mario Gulich”, Falda del Cañete, Argentina. Alemandri, V M., (ORCID: 0000-0002-3981-6600): Instituto Nacional de Tecnología Agropecuaria (INTA), Centro de Investigaciones Agropecuarias (CIAP), Instituto de Patología Vegetal (IPAVE), y Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), Unidad de Fitopatología y Modelización Agrícola (UFYMA), Córdoba, Argentina. Gómez Montenegro, B., (ORCID: 0009-0007-1667-5044): Instituto de Patología Vegetal (IPAVE), Centro de Investigaciones Agropecuarias (CIAP), Instituto Nacional de Tecnología Agropecuaria (INTA) y Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET), Unidad de Fitopatología y Modelización Agrícola (UFYMA), Córdoba, Argentina. Correspondencia a: pablopaccioretti@agro.unc.edu.ar
RESUMEN
Los datos climáticos derivados de imágenes o productos satelitales disponibles en la nube tienen gran cobertura en espacio y tiempo, buena precisión y, en general, son de libre acceso. Sin embargo, la obtención y descarga de variables climáticas a diferentes escalas se encuentra limitada por la falta de procedimientos computacionales estandarizados. El objetivo de este estudio fue desarrollar un código computacional que facilite el manejo de imágenes satelitales para obtención de variables climáticas en un dominio espacio-temporal. El producto ERA5 del servicio de Cambio Climático Copernicus fue usado como fuente de datos climáticos. El protocolo incluye la descarga desde la plataforma Google Earth Engine, con un código desarrollado en lenguaje R. Adiciona el preprocesamiento estadístico de los datos climáticos a escala quincenal y/o mensual. Combinando productos derivados de satélites con conocimiento agronómico sobre un cultivo, los datos climáticos pueden convertirse en variables biometeorológicas y usarse para el monitoreo espacio-temporal de cultivos. El proceso generado se validó superponiendo datos de variables biometeorológicas, en cada sitio de un estudio epidemiológico, sobre dos virus monitoreados por 15 años. El procedimiento puede aplicarse a otros productos satelitales que generan datos espaciales.
Palabras clave: Google Earth Engine, software R, ERA5, computación en la nube.
ABSTRACT
Climate variables data derived from satellite imagery or producís available in the cloud have wide coverage in space and time, good accuracy, and are generally freely accessible. However, obtaining and downloading climate variables at different spatial and temporal scales is limited by the lack of standardized computational procedures. The objective of this study was to develop a computational code to facilitate handling satellite images in order to derive climatic variables for a given spatiotemporal domain. The climate data was obtained from ERA5, a Copernicus Climate Change Service product. The protocol includes data download from the Google Earth Engine platform with a code developed in R language. The protocol developed includes statistical preprocessing of climatic data at fortnightly and/or monthly scale. By combining satellite-derived products with agronomic knowledge about a crop, climate data can be converted into biometeorological variables and used for spatiotemporal monitoring of crops. The process developed was validated by joint data from biometeorological variables at each site of an epidemiological study which has been monitoring two viruses for 15 years. The protocol may be applied to other satellite products using spatial data.
Keywords: Google Earth Engine, R software, ERA5, cloud computing.
INTRODUCCION
El clima es un factor empleado para explicar la distribución y desarrollo de organismos vivos y su relación con los recursos naturales que los rodean (Yim y Kira, 1975). Cuando las variables meteorológicas se relacionan con estados del cultivo, se obtienen indicadores o variables biometeorológicas (Gosling et al., 2014; Noce et al., 2020), del tipo precipitaciones o temperaturas previas a la siembra, durante el llenado de grano o la cosecha.
Enmarcado en el Servicio de Cambio Climático Copernicus (Copernicus Climate Change Service - C3S) de la Agencia Espacial Europea (ESA), el Centro Europeo para Previsiones Meteorológicas a Medio Plazo (European Centre for Medium-Range Weather Forecasts - ECMWF) está produciendo grandes conjuntos de datos climáticos a escala global. Se combinan modelos climáticos y datos observacionales de todo el mundo. Este producto es la quinta generación de Reanálisis
Europeo (European ReAnalysis - ERA5), sucesor de ERA-Interim (Muñoz-Sabater et al., 2021). Los productos ponen a disposición información para todo el mundo, a escala temporal diaria y mensual desde el año 1979. En consecuencia, cada vez es más factible mapear y modelar las variaciones o cambios espacio-temporales de las condiciones meteorológicas imperantes antes, durante y después de los cultivos anuales.
Gracias a la disponibilidad de datos climáticos masivos en diversas plataformas de Internet, es posible obtener información climática variada para un sitio georreferenciado sobre un territorio de interés. Actualmente, se cuenta con diferentes herramientas informáticas para la obtención de productos satelitales almacenados en la nube. Entre ellas, Google Earth Engine (GEE) (Gorelick et al., 2017) es una plataforma ampliamente utilizada que permite, a través de código de programación, la descarga y procesamiento de información proveniente de numerosas imágenes satelitales y datos geoespaciales desde la nube. Una ventaja de GEE, es que permite el procesamiento en la nube, esto es, por medio de Internet, pero por fuera del ordenador del usuario. A través de GEE, es posible preprocesar diferentes variables meteorológicas georreferenciadas a diferentes escalas, tanto espacial como temporal para su posterior descarga. Los datos pueden obtenerse para un sitio o lote agrícola, un país o, incluso, a escala planetaria, en diferentes períodos de tiempo.
La plataforma GEE permite la descarga de datos meteorológicos del producto ERA5, para cualquier sitio del planeta, desde el año 1979. Los datos se encuentran registrados por hora, lo que significa que pueden descargarse en intervalos horarios, o agregados en diferentes periodos de tiempo. Por ejemplo, promedio mensual o promedios mensuales calculados para cada hora del día. Considerando el creciente interés por análisis espacialmente explícitos, la obtención de variables biometeorológicas puede beneficiarse del cálculo de los productos climáticos, datos de estaciones meteorológicas georreferenciadas y a través de la interpolación basada en regresión o el reanálisis climático (Hu et al., 2016). Sin embargo, se realizaron limitados avances para automatizar la obtención y descarga de variables meteorológicas a diferentes escalas espaciales y temporales de la nube para su posterior correlación con el estado de los cultivos sometidos a diversas condiciones ambientales y la influencia de distintas enfermedades. El objetivo de este estudio fue desarrollar un protocolo computacional que facilitara la obtención, procesamiento, descarga y visualización de datos climáticos desde la nube para, de esta manera, obtener variables biometeorológicas que permitan el monitoreo de un evento particular asociado al desarrollo de un cultivo y la presencia/ausencia de virus. El protocolo propuesto integra, de manera novedosa, otras herramientas utilizadas para el procesamiento de datos espaciales. El uso de las herramientas desarrolladas en el software R y GEE facilita el accionar de futuros usuarios y sirve de base para otras implementaciones en el monitoreo de cultivos.
MATERIALES Y MÉTODOS
Datos
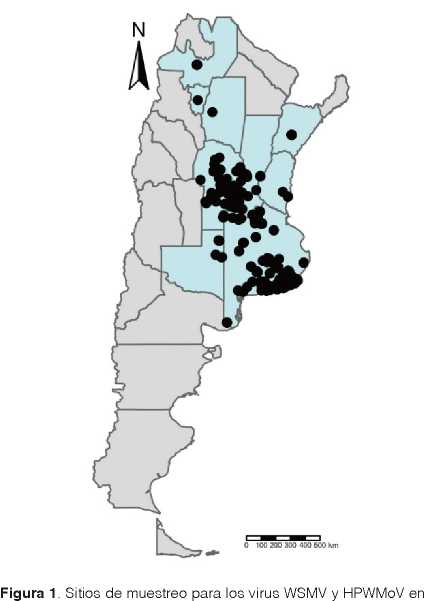
Utilizando la plataforma GEE, a través del paquete rgee (Aybar et al., 2020) del software R (R Core Team, 2021), se descargaron datos cada 14 días de temperatura, precipitaciones y temperatura de punto de rocío, para sitios con observaciones sobre la presencia de virus Wheat streak mosaic virus (WSMV) y High Plains wheat mosaic virus (HPWMoV) en trigo, correspondiente a quince años. De esta manera, la unidad observacional de la base de datos resultante se corresponde con la combinación de sitio y año, para los que se cuenta con información cada 14 días de cada variable biometeorológica en el periodo de interés. Las descargas se realizaron para 15 años, con datos de campo comprendidos en el período 2002-2021 (excepto los años 2008, 2016, 2017, 2019 y 2020). Durante esos años, se monitoreó la incidencia viral en 204 sitios de Argentina, acumulando 898 observaciones en el total de años muestreados. De cada muestra, se determinó la presencia/ausencia del virus por técnica serológica DAS-ELISA, con sueros comerciales específicos para cada virus. Ambos virus se transmiten por un ácaro eriófido, Aceria tosichella, por lo que la presencia de la enfermedad puede asociarse a las condiciones meteorológicas favorables para la diseminación de los ácaros vectores (Wosula et al., 2017). La región seleccionada para visualizar la variabilidad espacio-temporal de las variables climáticas está formada por 11 provincias de Argentina (Figura 1): La Pampa, Buenos Aires, Córdoba, Santa Fe, Entre Ríos, Santiago del Estero, Corrientes, Salta, Chaco, Tucumán y Jujuy, donde se realizó el monitoreo de la enfermedad. Los datos climáticos se obtuvieron del producto ERA5 Land (Muñoz-Sabater et al., 2021). Este producto contiene información climática de 50 variables (bandas), en una resolución espacial de 11132 x 11132 m. En este trabajo, se utilizaron datos de precipitaciones trigo. En color celeste, se resaltan las provincias muestreadas según reportes de enfermedad en metros, temperatura del aire y temperatura de rocío, ambos a dos metros de la superficie en grados Kelvin. Para una mejor interpretación de los valores, las precipitaciones se multiplicaron por 1000, para transformarlas a milímetros (mm) y las temperaturas se transformaron a grados centígrados (°C) restando 273,15 a cada valor. Para los muestreos realizados con relación al cultivo de trigo, la información biometeorológica descargada se obtuvo como el promedio, o acumulación en caso de precipitaciones de periodos de 14 días, durante los meses de abril, mayo y junio. La información espacial de los límites de las provincias muestreadas se obtuvo con Global Administrativa Unit Layars (GAUL), distribuido por FAO (Food and Agricultura Organization) de las Naciones Unidas. En la Figura 1 se evidencian los sitios de muestreo para los 15 años de estudio.

Protocolo de descarga y análisis
Mediante la interfaz de programación de aplicaciones (Application Programming Intarfaca - API), resulta posible utilizar y ejecutar código de GEE, desde clientes que utilizan JavaScript o Python. En el software de programación R (R Core Team, 2021), se desarrolló el paquete rgaa (Aybar et al., 2020) que permite el uso de la API de GEE de Python y la comunicación entre R y Python, que es posible usando el paquete raticulata (Ushey et al., 2022). La secuencia de código, resultado de este trabajo, permite la obtención de límites provinciales desde la nube; unión de archivos espaciales entre ocurrencia de virus y provincia de muestreo; mapeo de datos espaciales; descarga; obtención de medidas descriptivas de las imágenes descargadas y visualización de datos climáticos temporales, para cada sitio de muestreo y para la región en estudio. En el siguiente enlace: https://github.com/PPaccioretti/descarga_datos_ climaticos se encuentra disponible el script y los datos para reproducir el análisis.
Además de permitir la descarga de una de las capas de las variables meteorológicas disponibles en ERA5 para un determinado dominio espacial y periodo de tiempo, el protocolo incluye herramientas de preprocesamiento que facilitan el manejo de datos tipo ráster, utilizando el paquete stars (Pebesma, 2022) y la visualización de los datos. Es posible generar mapas de variabilidad espacial de las variables meteorológicas, mediante el uso del paquete tmap (Tennekes, 2018) y ggplot2 (Wickham, 2016). Se describe la variabilidad espacio-temporal, concatenando la variabilidad espacial de una serie de imágenes temporales. Se calculan promedios o acumulaciones de valores de estas variables para distintos periodos o semanas relacionadas con el ciclo del cultivo. Finalmente, para visualizar el impacto de las variables biometeorológicas seleccionadas, se realizan gráficas mediante el paquete ggplot2 (Wickham, 2016) en R. Estas gráficas permiten contrastar las medias de las variables climáticas entre sitios con presencia vs. sitios con ausencia del evento de monitoreo. En el caso de ilustración, el evento monitoreado es la presencia del virus bajo estudio.
Por último, se analizó la presencia y ausencia de cada virus en relación con valores de las variables biometerológicas resumidas en periodos de 14 días para todo el otoño (siete periodos). La relación fue estudiada estadísticamente, mediante un modelo lineal generalizado para una variable binaria (presencia/ausencia del virus en el cultivo) y 21 variables regresoras, representadas por la temperatura promedio, precipitación acumulada y punto de rocío promedio, para cada uno de los siete períodos en estudio. El modelo fue ajustado con enlace canónico, utilizando la función glm del softwara R (R Core Team, 2021).
RESULTADOS Y DISCUSIÓN
Visualización de variabilidad espacial de datos climáticos
En la Figura 2a, se muestra la temperatura promedio para los meses de abril, mayo y junio, en los años donde se realizaron muestreos. El promedio de temperatura para toda la región fue de 15,7 °C. El valor máximo promedio observado en los años en estudio fue de 23,8 °C. El valor mínimo promedio fue de -10 °C, observado en la zona noroeste de la región. Las precipitaciones acumuladas promedio (Figura 2b) para la región en estudio fueron de 190 mm para otoño, variando entre 5 mm y 954 mm. La temperatura de punto de rocío promedio fue de 8,9 °C (Figura 2c). En el noroeste de la región, se observaron las menores temperaturas de punto de rocío (-26.4 °C) y la mayor altitud (Figura 2d).
Visualización de variabilidad espacio temporal en datos climáticos
En la Figura 3, se visualizan las precipitaciones acumuladas de la región para los años en estudio. En el año 2009, se observaron las menores precipitaciones promedio (98 mm), mientras que, en el año 2002, se observó la mayor precipitación promedio 278 mm). Las temperaturas promedio se encontraron entre 15 °C y 17,1 °C. El año con promedio de temperatura más bajo fue el 2007 y el año con mayor temperatura promedio fue el 2009.
Relación datos bioclimáticos con presencia de enfermedad
La presencia/ausencia del virus HPWMoV, en relación con valores de las variables biometeorológicas resumidas para períodos de 14 días desde el comienzo del otoño (semana 0), se sustentó estadísticamente con el ajuste de un modelo lineal generalizado. Los resultados Indicaron que una variable de mayor impacto es la precipitación acumulada de las semanas 7 y 8 (relación negativa con p < 0,0001) y de las semanas 11-12 y 13-14 (relación positiva, p = 0,0001 y p < 0,0001, respectivamente) (Figura 4). El impacto de las precipitaciones también se observó sobre la presencia del virus WSMV, que fue mayor cuando ocurrieron mayores precipitaciones durante la semana 5 y 6 (p < 0,0001) y menor con precipitaciones altas hacia finales del otoño (p < 0,0001) (Figura 5).
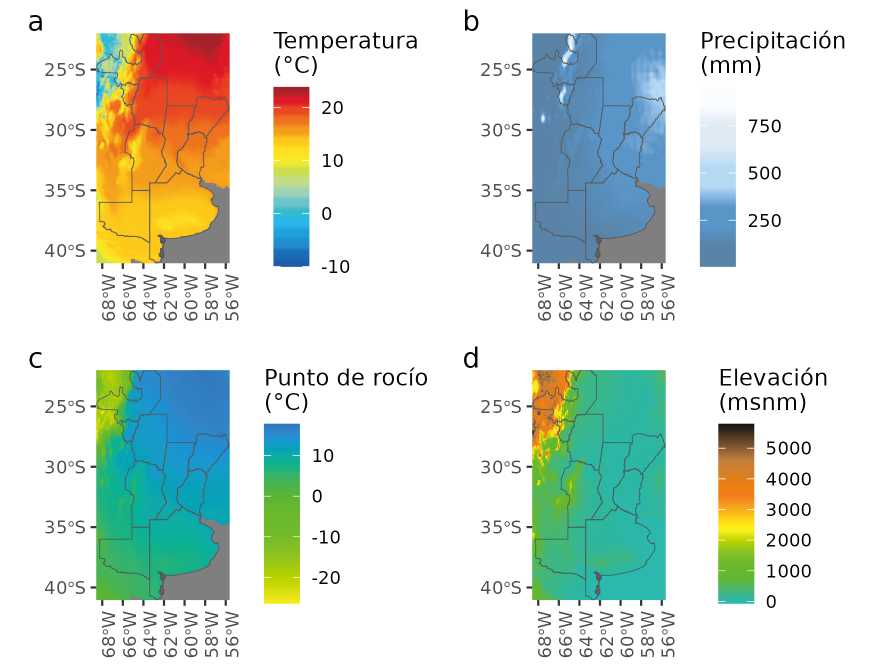
Figura 2: Mapa de variabilidad espacial de: a) temperatura promedio, b) precipitaciones acumuladas promedio, c) temperatura de punto de rocío promedio, calculados utilizando el período de abril, mayo y junio de la región en estudio en los años donde se realizó el muestreo y d) elevación de la región en estudio.
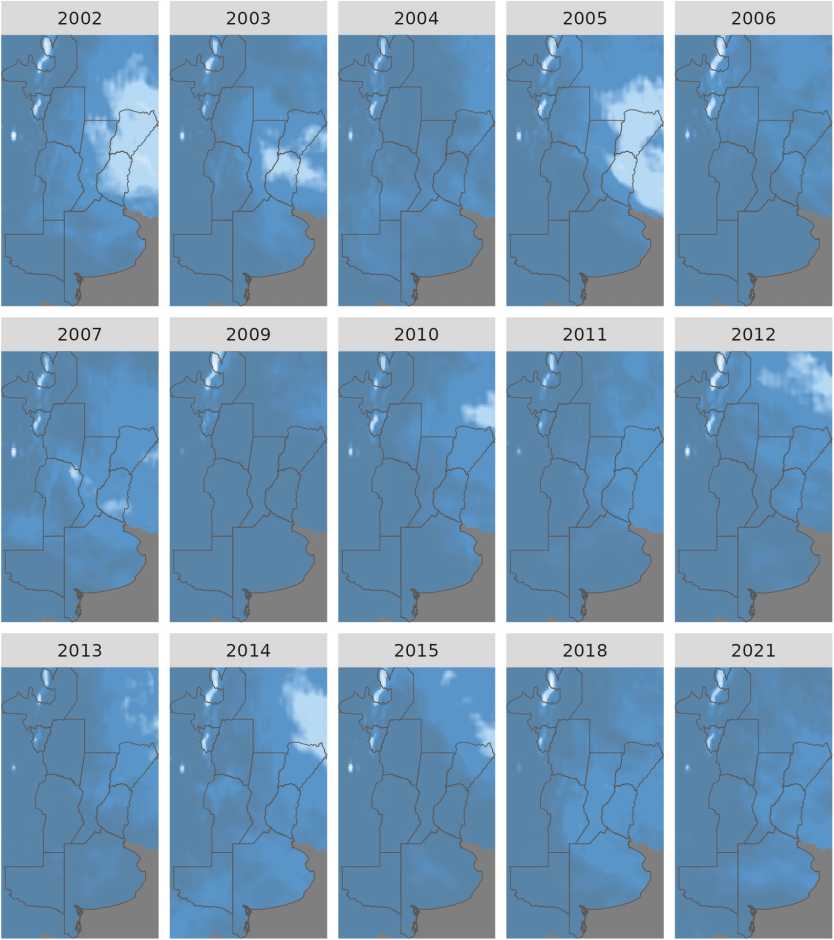
Figura 3: Variabilidad espaciotemporal de precipitaciones acumuladas de abril, mayo y junio en los años muestreados en la región de estudio.
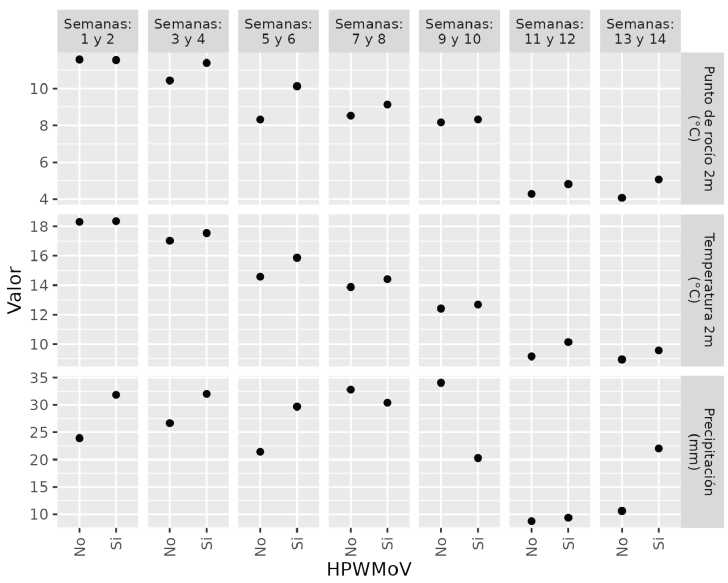
Figura 4: Relación entre presencia/ausencia del virus HPWMoV en cultivo de trigo, en función del valor de punto de rocío promedio, temperatura promedio y precipitaciones acumuladas promedio, estimadas cada dos semanas durante el otoño.
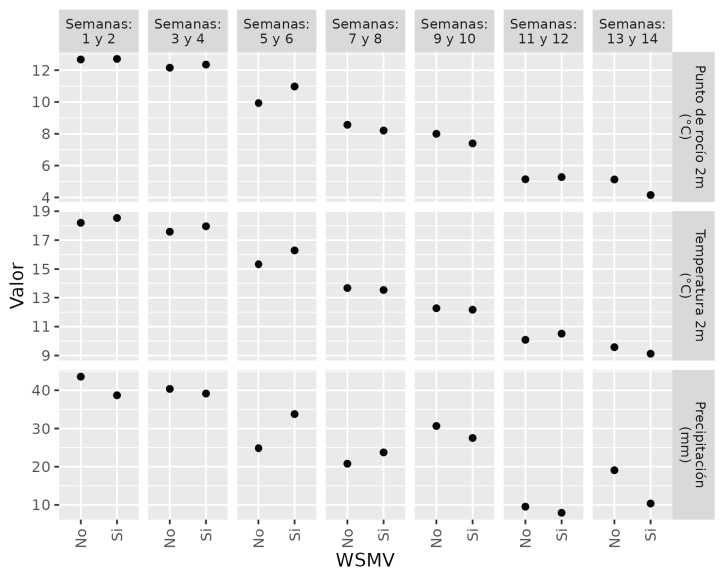
Figura 5: Relación entre presencia/ausencia del virus WSMV en trigo, en función del valor de punto de rocío promedio, temperatura promedio y precipitaciones acumuladas promedio de las variables biometeorológicas, obtenidas cada 14 días desde el comienzo del otoño.
CONCLUSIÓN
El protocolo estadístlco-computaclonal desarrollado vlablllza la descarga de variables climáticas desde la nube en diferentes escalas espaciales y temporales. Además, facilita el análisis exploratorlo del lmpacto del cllma sobre el evento de interés. Se implementó exitosamente para descargar datos de precipitación, temperatura y punto de rocío, que fueron relacionados con la presencia/ausencia de virosis en trigo. El script desarrollado puede utilizarse para el estudio de otros eventos de cultivo con distribución binarla (presencia/ausencia) que hayan sido georreferenclados.
AGRADECIMIENTOS
Las investigaciones que sustentan este estudio fueron posibles gracias al Proyecto de Unidad Ejecutora (PUE) de la Unidad de Fitopatología y Modelización Agrícola (UFYMA), dependiente del Instituto Nacional de Tecnología Agropecuaria (INTA) y del Consejo Nacional de Investigaciones Científicas y Técnicas (CONICET). Además, se agradece a la Dra. Vanlna Alemandrl por la base de datos utilizada para la ilustración.
Fecha de recepción: 06/12/2022;
fecha de aceptación: 05/06/2023
BIBLIOGRAFÍA
Aybar, C., Wu, Q., Bautista, L., Yali, R. y Barja, A. (2020). rgee: An R package for interacting with Google Earth Engine. Journalof Open Source Software, 5(51), 2272. https://doi.ora/10.21105/ioss.02272 Gorelick, N., Hancher, M., Dixon, M., Ilyushchenko, S., Thau, D. y Moore, R. (2017). Google Earth Engine: Planetary-scale geospatial analysis for everyone. Remote Sensing of Environment, 202, 18-27. https:// doi.org/10.1016/j.rse.2017.06.031 Gosling, S. N., Bryce, E. K., Dixon, P. G., Gabriel, K. M. A., Gosling, E. Y., Hanes, J. M., Hondula, D. M., Liang, L., Bustos Mac Lean, P. A., Muthers, S., Nascimento, S. T., Petralli, M., Vanos, J. K. y Wanka, E. R. (2014).
A glossary for biometeorology. International Journal of Biometeorology, 58(2), 277-308. https://doi.org/10.1007/s00484-013-0729-9
Hu, Z., Hu, Q., Zhang, C., Chen, X. y Li, Q. (2016). Evaluation of reanalysis, spatially interpolated and satellite remotely sensed precipitation data sets in central Asia. Journal of Geophysical Research: Atmospheres, 121(10), 5648-5663. https://doi.org/10.1002/2016JD024781
Muñoz-Sabater, J., Dutra, E., Agustí-Panareda, A., Albergel, C., Arduini, G., Balsamo, G., Boussetta, S., Choulga, M., Harrigan, S., Hersbach, H., Martens, B., Miralles, D. G., Piles, M., Rodríguez-Fernández, N. J., Zsoter, E., Buontempo, C. y Thépaut, J.-N. (2021). ERA5-Land: a state-of-the-art global reanalysis dataset for land applications. Earth System Science Data, 13(9), 4349-4383. https://doi.org/10.5194/essd-13-4349-2021
Noce, S., Caporaso, L. y Santini, M. (2020). A new global dataset of bioclimatic indicators. Sclentlfic Data, 7(1), 398. https://doi.org/10.1038/s41597-020-00726-5
Pebesma, E. (2022). stars: Spatiotemporal Arrays, Raster and Vector Data Cubes.
R Core Team. (2021). R: A Language and Environment for Statistical Computing (4.1.2). https://www.r-project. org/Tennekes, M. (2018). tmap : Thematic Maps in R. Journal of Statistical Software, 84(6). https://doi.org/10.18637/ jss.v084.i06
Ushey, K., Allaire, J. J. y Tang, Y. (2022). reticulate: Interface to “Python”.
Wickham, H. (2016). ggplot2: Elegant Graphics for Data Analysis. Springer-Verlag New York.
Wosula, E. N., Tatineni, S., Wegulo, S. N. y Hein, G. L. (2017). Effect of temperatura on wheat streak mosaic disease development in winter wheat. Plant Disease, 101(2), 324-330. https://doi.org/10.1094/PDIS-07-16-1053-RE
Yim, Y. y Kira, T. (1975). Distribution of forest vegetation and climate in the Korean península: I. distribution of some indices of thermal climate. Japanese Journal of Ecology, 25(2), 77-88. https://doi.org/10.18960/ seitai.25.2 77